Rを4.2系にバージョンアップしたらRstanの導入でつまずいた話
今回はRにRstan(Rで利用可能なStan)を導入しようとしてつまずいた時の備忘録です。(OSはWindowsです)
Rのバージョンを”うっかり”4.2系にバージョンアップしたために、Rstanの導入に苦労しました。
R4.2系のままRstanを導入する際の助けになればと思います。
(Rの操作はRStudioから行います)
StanとRstan
#「Stan」は統計的推論のためのプラットフォームです。
ベイズ統計モデルの解析などに利用されます。
「Rstan」はRからStanを呼び出せるようにしたもので、R言語と統合されています。
他に有名なものに「PyStan」があります。PyStanはPythonからStanを利用するためのモジュールです。
Rの最新版(4.2.1)を取得する
#リリースされているRのバージョンを確認します。
Rの最新版を確認したところ、2022年06月23日にバージョン4.2.1がリリースされていました。
今回の検証用PCにはバージョン4.2.1をインストールすることにしました。
RStudio
#RStudioのバージョンも確認します。
今回は以下のバージョンを使用することにしました。
こちらも2022年5月20日にリリースされた比較的新しいバージョンです。
RStudio 2022.02.3+492 "Prairie Trillium" Release (1db809b8323ba0a87c148d16eb84efe39a8e7785, 2022-05-20) for Windows
最初の注意点(Rtoolsのバージョン)
#Rstanを使用するためにはRtoolsの導入が必要とのことなのでRtoolsをインストールします。
しかし、RtoolsはRのバージョンに対して制約があります。
制約は以下のようになっていました。
現環境のRのバージョンが4.2.1なので、Rtoolsは4.2版を使用する必要があります。
筆者は何も考えずに4.0版をダウンロードしてしまい、再度4.2版をダウンロードする羽目になりました。
お気を付けください。
ダウンロードには多少時間を要します。
ダウンロードが出来たらインストーラを起動し、デフォルト設定のままインストールします。
Rstanのインストール
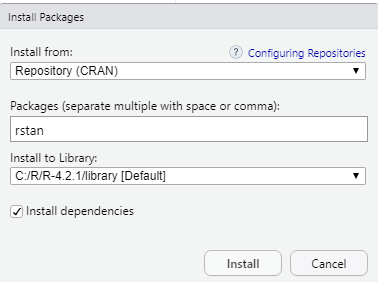
#RStudioを起動し、パッケージインストーラから「Rstan」をインストールします。
インストールを開始すると関連パッケージが大量にダウンロードされます。
少し時間がかかりますが、インストールが終了するまで待ちます。
Rstanライブラリの読み込み
#Rstanのインストールが完了したら、RStudio上で以下のコマンドを実行しRstanライブラリを読み込みます。
library(rstan)
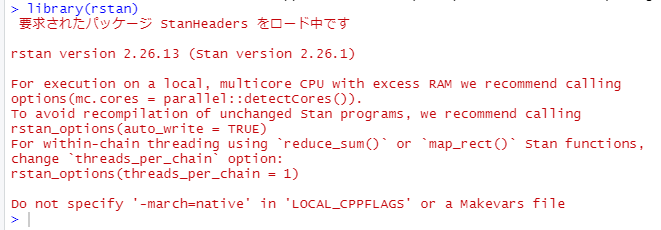
以下のようなメッセージが表示されました。
(Rのメッセージはエラーでなくても赤字だったりするので毎回ビクっとします)
Rstanのバージョンが「2.21.5」であることがわかります。
マルチコアCPUの場合が何だかんだと言われていますが、取りあえず導入できているようなので先に進みます。
Rstanサンプル実行(エラー)
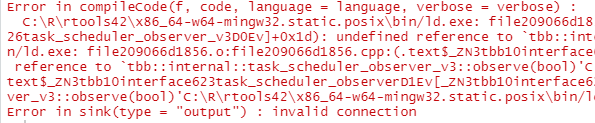
#適当なRstanサンプルを実行しようとしたところ、以下のエラーが発生しました。
まったく思い当たる節がありません。
Rのバージョンを4.2.0にダウングレードしたり、Rstanを再度インストールしたり、諸々実施しましたがエラーは解消されせんでした。
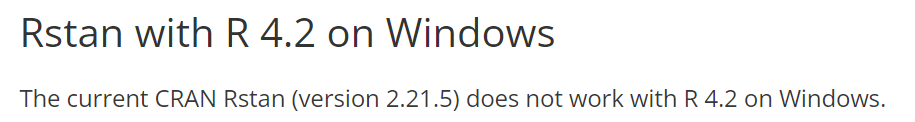
Rのバージョンを4.1.3にダウングレードするしか方法が無いかと途方に暮れていた時に、ネット上に次のような情報を見つけました。
え?!
カレントバージョン2.21.5のRstanがR4.2では動作しない!
それもWindows限定!
どうやらRのバージョンを4.2系にバージョンアップしたことが仇となってしまったようです。
強制的にRstanのバージョンを変更する
#幸いなことに先ほど閲覧したサイトに対応方法が載っていたました。
要は
- 古いバージョンのRstanをアンインストールして
- Rstanリポジトリからパッケージを取得せよ
のようです。
以下を実行します。
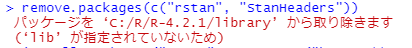
RstanとStanHeadersパッケージの削除
remove.packages(c("rstan", "StanHeaders"))
Rstanリポジトリからパッケージを取得
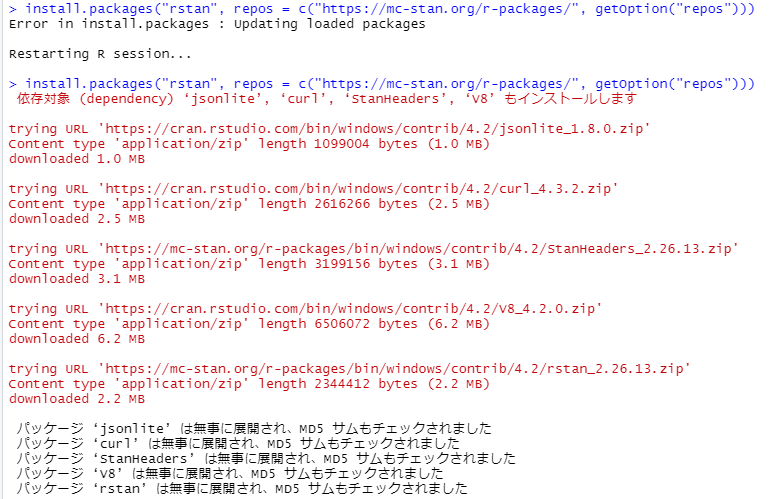
install.packages("rstan", repos = c("https://mc-stan.org/r-packages/", getOption("repos")))
パッケージがインストールできたようです。
Rstanのバージョンを確認します。
Rstanのバージョンが「2.26.13」に更新されています。
別のPCでRを実行していて判明したのですが、Rstan 2.26.13 はRのバージョン4.2.1のもとで作成されたらしく、Rのバージョン4.2.0の環境で使用すると「バージョンが合っていない」旨のワーニングが表示されました。
Rのバージョンは4.2.1にした方が良いでしょう。
Rstanサンプル実行(成功)
#Rstanの動作を確かめるために簡単なサンプルを実行させます。
サンプルは「コインを10回投げて80%の確率で表が出る二項分布」の計算です。
Rstanファイル「coin.stan」を以下のように記述します。
data {
int N;
int n;
int x[n];
}
parameters {
real<lower=0, upper=1> p;
}
model {
x ~ binomial(N, p);
}
実行用Rファイルを以下のように記述します。
# Rstanライブラリの読み込み
library(rstan)
# オプション設定(計算の並列化)
rstan_options(auto_write=TRUE)
options(mc.cores=parallel::detectCores())
# コインを10回投げて、80%の確率で表が出るデータを作成する
x <- rbinom(n = 10, size = 1, prob = 0.8)
data <- list(N = 1, n = length(x), x = x)
# Stan実行
fit <- stan(file = 'coin.stan', data = data)
# 結果出力
fit
# 結果表示
stan_hist(fit)
実行結果が以下のように出力されました。
平均(期待値)は0.75となっており、サンプルデータに設定した0.8に近い値になっています。
グラフも出力されました。
イメージ通りのグラフの形になっています。
正しくRstanを実行できたようです。
まとめ
#今回はRのバージョンを最新版4.2.1にバージョンアップしてしまったために、余計な手間がかかってしまいました。
最新版を試すときは慎重に実施しないといけないと再確認しました。(リリースノートはちゃんと読もう)
Node.jsのNVMやPythonのCondaように開発環境のバージョン自体を簡単に切り替えられる仕組みがRにも有ったらなぁ、と思う今日この頃です。
皆様もお気を付けください。
データ分析に活用して頂ければ幸いです。